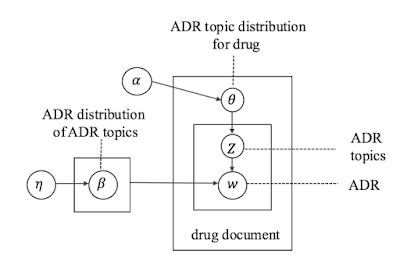
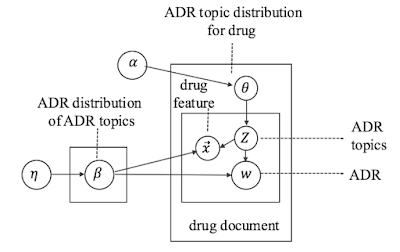
概要
LDAの結果を可視化するツールLDAvisを使ってモデルを俯瞰する。
モデルの構築
モデルを構築するRスクリプトをリスト1に示す。
################################################
#
# モデルの生成
#
################################################
# log likelihood の最大値を与えるトピック数=350
topic_num = 350
burnin = 500
iter = 1000
keep = 10
model <- LDA(dpc_dtm, k = topic_num, method = "Gibbs", control = list(burnin = burnin, iter = iter, keep = keep) )
model
LL <- data.frame(
topic_nums = seq(10, 1500, 10),
logLiks = model@logLiks
)
################################################
#
# 対数尤度の変化
#
################################################
ggplot(LL, aes(x=topic_nums, y=logLiks)) + geom_line() + labs(title="Evolution of log likelihood along with iterations", x="iterations",y="log likelihood") + theme(plot.title=element_text(vjust=0.5, hjust=0.5))
リスト1.モデルの構築
モデルの構築にあたってGibbsサンプリングを用い、トピック数は350とした。これはブログ記事「
topicmodelsパッケージでLDA(パーツ作りました)」の図4で、トピック数が350のとき対数尤度の調和平均が最大になったことに基づいている。つまり、モデル選択の基準として尤度を最大にするという基準を用いている。
また、LDAのオプションであるburninやiterの妥当性を確かめるためにiterationが10, 20, ..., 1500に対する対数尤度を求めてプロットしたのが図1である。
 |
| 図1.対数尤度のiteration依存性 |
図1から、対数尤度はiterationsが200あたりで急激な増加から緩やかな増加に転じており、500を超えたあたりではほぼ飽和しているとみなせるのでburnin=500, iter=1000という設定は妥当なものと考えられる。
LDAvisの結果
作成したモデルをLDAvisを使って可視化するスクリプトをリスト2に示す。
################################################
#
# LDAvisで使うデータを作成する関数
# fit: LDAモデル
# doc_term: DocumentTermMatrix
#
################################################
buildVisdata1 <- function(fit, doc_term) {
phi <- posterior(fit)$terms %>% as.matrix
theta <- posterior(fit)$topics %>% as.matrix
vocab <- colnames(phi)
doc.length <- rowSums(as.matrix(doc_term))
term.frequency <- colSums(as.matrix(doc_term))
params <- list(phi = phi,
theta = theta,
doc.length = doc.length,
vocab = vocab,
term.frequency = term.frequency)
return(params)
}
install.packages("tsne")
library("tsne")
svd_tsne <- function(x) tsne(svd(x)$u)
install.packages('servr')
library(servr)
# LDAvis
install.packages("LDAvis")
library(LDAvis)
VisualizeModel <- function(fit, doc_term, dir) {
params <- buildVisdata1(fit, doc_term)
json <- createJSON(phi = params$phi,
theta = params$theta,
doc.length = params$doc.length,
vocab = params$vocab,
term.frequency = params$term.frequency,
mds.method = svd_tsne
)
serVis(json, out.dir = dir, open.browser = TRUE)
}
VisualizeModel(model, dpc_dtm, './')
リスト2.
リスト2を実行した結果を図2に示す。
 |
| 図2.LDAvisの実行結果(トピック数=350) |
左図はトピックの全体像を示す。各円がトピックを表し、その面積はコーパス中におけるそのトピックの相対的な割合(prevalence)に比例して描かれている。各円に付けられた数字は割合の大きい順につけられた番号である。
右図は選択されたトピックに関連のある単語を横棒グラフで示したものである。灰色の棒はコーパス全体における各単語の出現頻度を表しており、赤色の棒は選択されているトピックに固有な出現頻度を表している。
図2で問題なのは
- 右図で単語(疾患)がコードで表されており、疾患名がわからない
- トピックに関連する疾患がたった1つしかないように見える
点である。
まず1については、buildVisdata1でvocabへcolnames(phi)、すなわちposterior(fit)$termsのカラム名を代入しているので、疾患名ではなくDPCの疾患コードが設定され、当然の結果になっている。
ここに疾患名を出すのであれば、buildVisdata1でvocabへ疾患名リストを設定する必要がある。疾患名リストが疾患コード(DPCコード)と対応付けられていればそれを使えばよい。
まず、現在のvocabには
colnames(posterior(model)$terms)
が設定されている。
次に、疾患名と疾患コードの関係は、dpc_tibbleから(DPC2, 疾患名)を抽出してDPC2で圧縮すればよい。最後にDPC2をキーとして結合し、疾患名だけ抜けば疾患名リストが完成する。以上の処理をリスト3に示す。
disease_dic_tibble <- dpc_tibble %>%
tidyr::unite(疾患手術名, c('疾患名', '手術')) %>%
dplyr::select(DPC2, 疾患手術名) %>%
dplyr::distinct(DPC2,.keep_all=TRUE) %>%
dplyr::arrange(DPC2)
terms_tibble <- tbl_df(colnames(posterior(model)$terms))
colnames(terms_tibble) <- c("DPC2")
vocab_tibble <- dplyr::left_join(terms_tibble, disease_dic_tibble, by="DPC2")
disease_names <- vocab_tibble$疾患手術名
リスト3.疾患名リストの作成
こうして求めた疾患名リスト「disease_names」をリスト2のbuildVisdata1内でvocabへ設定して実行してみた。すると何も表示されない。そこで、VisualizeModelが作成したJSONデータ「lda.json」を見てみると、文字コードがShift_JISになっている。
そこで、index.htmlのcharsetをutf-8からShift_JISに変更してみた。すると今度は”d3がない”といった関係のないエラーが出る。どうも、Shift_JISではd3(グラフィックスライブラリ)はだめなようである。ということは、CSVファイルから作り直さなければならないということらしい。
WindowsのR-Studioは
デフォルトでShift-JISになっているので、このあたりから
設定しなおす必要がある(ああ大変だ・・・Pythonではこんな面倒はなかったのに・・・日本語が絡むと絶対に何か起きる)。
そこで、その場しのぎで「lda.json」の文字コードを手作業でShift-JISからutf-8に変換してみた。その結果を図2bに示す。
 |
| 図2b.疾患名を表示したLDAvis |
これは下記のURLから操作できる。
http://hinfokumw.html.xdomain.jp/lda/LDAvis/350/
トピックに含まれる疾患の数
まず、トピックごとの疾患の出現確率
φを抽出する。それを行うのがposterior(model)$termsである。リスト4は各トピックがどのくらいの疾患を含んでいるか調べるスクリプトである。
phi <- posterior(model)$terms
threshold <- 1 / model@wordassignments$ncol # 1/V
terms_per_topic <- unlist(
lapply(1:model@k, function(k) {
return(sum(phi[k,] > threshold))
})
)
df <- data.frame(value = terms_per_topic)
ggplot(df, aes(x = value)) +
geom_histogram(binwidth = 1) +
annotate("text", x=25, y=40, label=paste("k =", model@k), hjust=0) +
annotate("text", x=25, y=38, label=paste("トピックに含まれる最大単語数 =", round(max(terms_per_topic), digit=1)), hjust=0) +
annotate("text", x=25, y=36, label=paste("トピックに含まれる最低単語数 =", round(min(terms_per_topic), digit=1)), hjust=0) +
annotate("text", x=25, y=34, label=paste("トピックに含まれる平均単語数 =", round(mean(terms_per_topic), digit=1)), hjust=0) +
annotate("text", x=25, y=32, label=paste("threshold = 1 / ", model@wordassignments$ncol), hjust=0) +
labs(title = "トピックを構成する単語(疾患)の数", x = "単語の数", y = "頻度")
リスト4.トピックに含まれる疾患数の可視化スクリプト
posterior(model)$termsはトピック数×疾患数のmatrixで、(
k,
v)要素にはトピック
kに含まれる疾患
vの割合(φ
kv)が格納されている。
φ
kvの閾値thresholdとして1 / model@wordassignments$ncolを設定する。ここで、model@wordassignments$ncolには疾患数として801という値が入っているので、閾値はthreshold=1/801=0.00125になる。
上記のトピック×疾患matrixの要素がこの閾値を超えるものだけをそのトピックの構成疾患とみなして、各トピックがいくつの疾患で構成されているかを調べる。それが、terms_per_topicで、添え字がトピック番号、要素の値がそのトピックを構成する疾患数になっている。こうして求めたterms_per_topicをもとにヒストグラムを描いたのが図3である。
 |
| 図3.トピックに含まれる疾患数 |
トピックに含まれる平均疾患数は12疾患で(中央値:7、最頻値:5)図に示すようにロングテールの分布を示す。
病院のトピック分布
同様にして各病院がいくつのトピックから構成されているかを求めてヒストグラムにするスクリプトをリスト5に、その結果を図4に示す。
theta <- posterior(model)$topics
threshold <- 1 / model@k
topics_per_document <- unlist(
lapply(1:model@wordassignments$nrow, function(d) {
return(sum(theta[d,] > threshold))
})
)
df <- data.frame(value = topics_per_document)
ggplot(df, aes(x = value)) +
geom_histogram(binwidth = 1) +
annotate("text", x=25, y=40, label=paste("k =", model@k), hjust=0) +
annotate("text", x=25, y=38, label=paste("病院が含む最小トピック数 =", round(min(topics_per_document), digit=1)), hjust=0) +
annotate("text", x=25, y=36, label=paste("病院が含む最大トピック数 =", round(max(topics_per_document), digit=1)), hjust=0) +
annotate("text", x=25, y=34, label=paste("病院が含む平均トピック数 =", round(mean(topics_per_document), digit=1)), hjust=0) +
annotate("text", x=25, y=32, label=paste("threshold = 1 / ", model@k), hjust=0) +
labs(title = "病院に含まれるトピックの数", x = "トピックの数", y = "病院の数")
リスト5.病院のトピック分布を求めてヒストグラムにするスクリプト
閾値は1 / model@kとした。ここで、model@kはトピック数(350)である。
 |
| 図4.病院のトピック分布 |
図4に示すように、病院が含むトピック数の最大は115で最小は3、平均は57である(中央値も57)。
図4から次のことがわかる。
- 図4はほぼ左右対称である
- 最大トピック数は350であるが、最も多いトピックを含む病院でも115トピックしか含んでいない
トピックを特徴づける疾患
各トピックに含まれる疾患の割合が閾値(1 / model@wordassignments$nrow=1/1664)を超えるものについて、その割合の降順に並べたリストを作成するスクリプトをリスト6に示す。
install.packages("radiant.data")
library(radiant.data)
phi_tibble <- dplyr::as_tibble(posterior(model)$terms, rownames=NA) %>%
radiant.data::rownames_to_column('topic') %>%
tidyr::gather(key = DPC2, value = phi, -topic)
phi_tibble$topic <- as.integer(phi_tibble$topic)
phi_with_disease_name <- phi_tibble %>%
dplyr::filter(phi > 1 / model@wordassignments$nrow) %>%
dplyr::arrange(topic, desc(phi)) %>%
dplyr::left_join(disease_dic_tibble, by = "DPC2")
write.csv(phi_with_disease_name, "./phi_with_disease_name.csv")
リスト6.トピックに含まれる疾患をリストアップするスクリプト
トピック×疾患マトリックスposterior(model)$termsをtibble型に変換するためにas_tibbleを使っている。これは、tbl_dfだと行名が失われるからである。as_tibbleを使うと、rownames=NAとすることにより、行名を残すことができる(rownamesを省略すると行名は除去される)。
tibble型に変換したらrownames_to_columnによって行名を列に変換し(列名は引数に指定した'topic')、gatherによってデータフレームの構造を"列型"から"行型"に変換する。ここで"列型"というのは、データが列方向に伸びているデータ構造のことで、列をバラバラに切り離して行方向にデータを並べたものを"行型"と呼んでいる。ちょうどリレーショナルデータベースにおいて繰り返しを取り除く第一正規化に似た処理である。
gatherの第一引数keyには”列型”の列名を値とする項目に付ける名前を指定する。ここではDPC2としている。第2引数valueには列名に対応する値に付ける名前を指定する。ここではphiとしている。最後の引数-topicは”列型”のデータフレームの列
topic以外すべての列をバラバラにすることを意味している(図5)。
 |
| 図5.dplyr::gather()関数のイメージ |
リスト7にはリスト6の実行結果を示す。
> phi_with_disease_name
# A tibble: 5,173 x 4
topic DPC2 phi 疾患手術名
<int> <chr> <dbl> <chr>
1 1 05017002 0.631 閉塞性動脈疾患_02
2 1 05013099 0.323 心不全_99
3 1 04008099 0.0150 肺炎等_99
4 1 06003501 0.00226 結腸(虫垂を含む。)の悪性腫瘍_01
5 1 06016001 0.00192 鼠径ヘルニア_01
6 1 05005099 0.00140 狭心症、慢性虚血性心疾患_99
7 1 06002097 0.000881 胃の悪性腫瘍_97
8 2 09001002 0.553 乳房の悪性腫瘍_02
9 2 09001005 0.333 乳房の悪性腫瘍_05
10 2 06003501 0.0559 結腸(虫垂を含む。)の悪性腫瘍_01
# ... with 5,163 more rows
>
リスト7.各トピックを特徴づける疾患